Imagerie de la maladie d'Alzheimer dans les essais cliniques Vue d'ensemble
L'imagerie multimodale est largement utilisée dans les essais cliniques de phase précoce et tardive de la maladie d'Alzheimer (MA). Les principales utilisations de l'imagerie sont les suivantes :
- IRM
- Lectures d'éligibilité
- Lectures de sécurité, y compris ARIA-E et ARIA-H
- Mesures quantitatives des critères d'efficacité (par exemple, IRMvolumétrique )
- TEP
- Éligibilité et enrichissement de la population étudiée
- Mesures quantitatives des critères d'évaluation de l'efficacité (par exemple, SUVR , étendue du cerveau)
L'utilisation efficace de la neuro-imagerie dans les essais cliniques sur la maladie d'Alzheimer nécessite la combinaison harmonieuse d'opérations d'étude rigoureuses, de lectures radiologiques avec un suivi de données fiable et de logiciels de traitement et d'analyse d'images validés et à la pointe de la technologie pour obtenir des mesures quantitatives à partir d'images de haute qualité.
Biomarqueurs d'imagerie pour la maladie d'Alzheimer
-
- IRM volumétrique (volumes régionaux et épaisseur corticale)
- TEP amyloïde
- Tau TEP
- FDG TEP
- IRM de perfusion ASL
- iRMf (état de repos et en fonction de la tâche)
- Imagerie par diffusion(par exemple, DTI, NODDI, imagerie de l'eau libre)
Points forts de nos recherches sur l'imagerie de la maladie d'Alzheimer
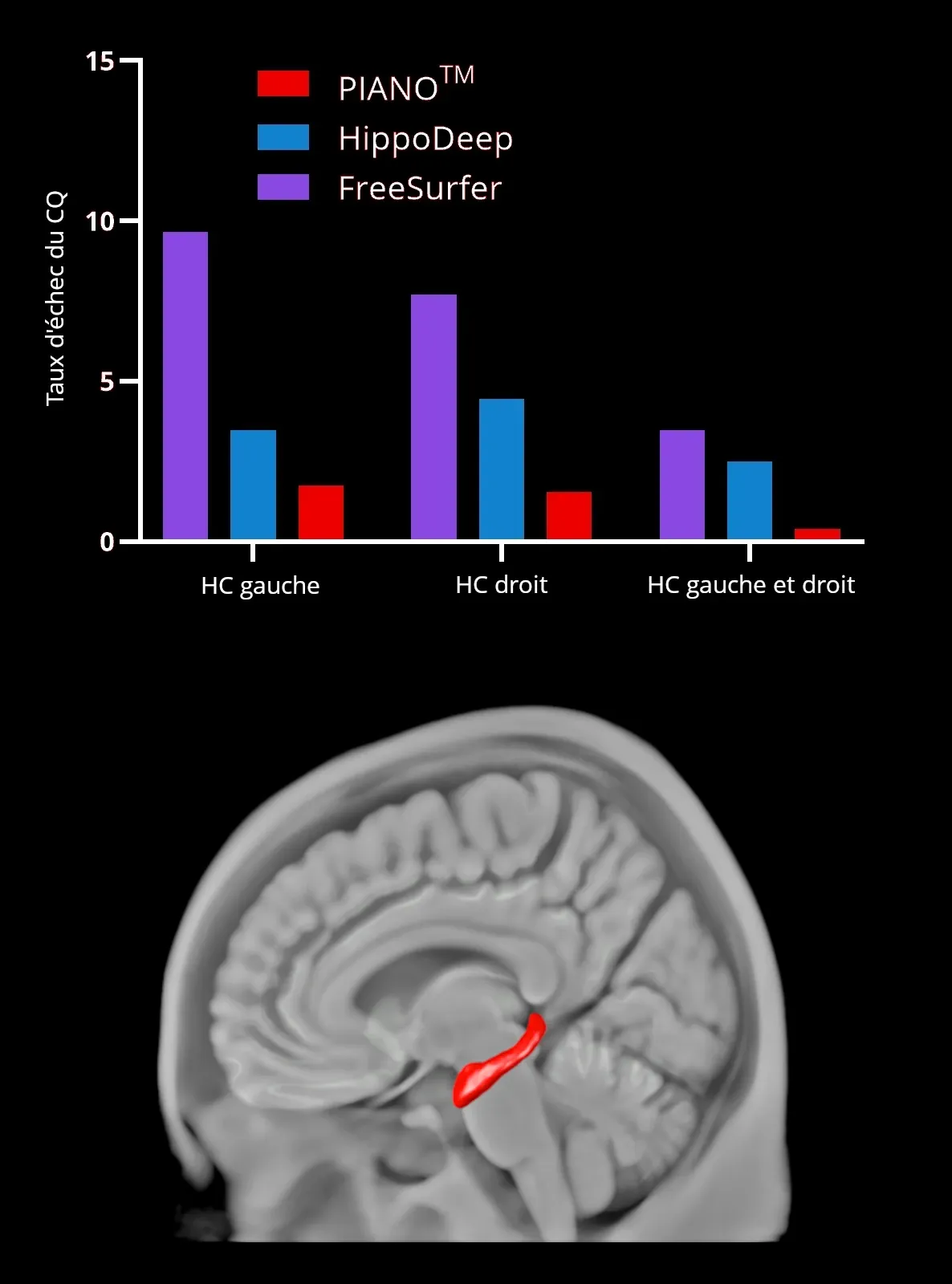
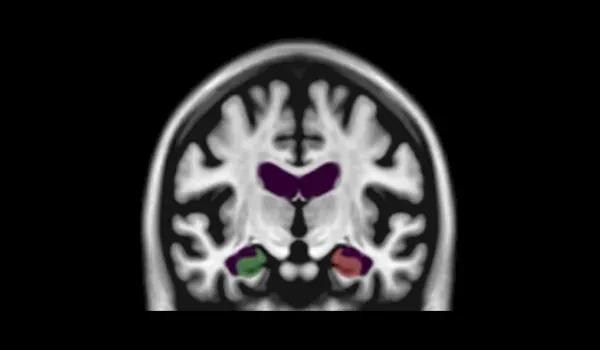
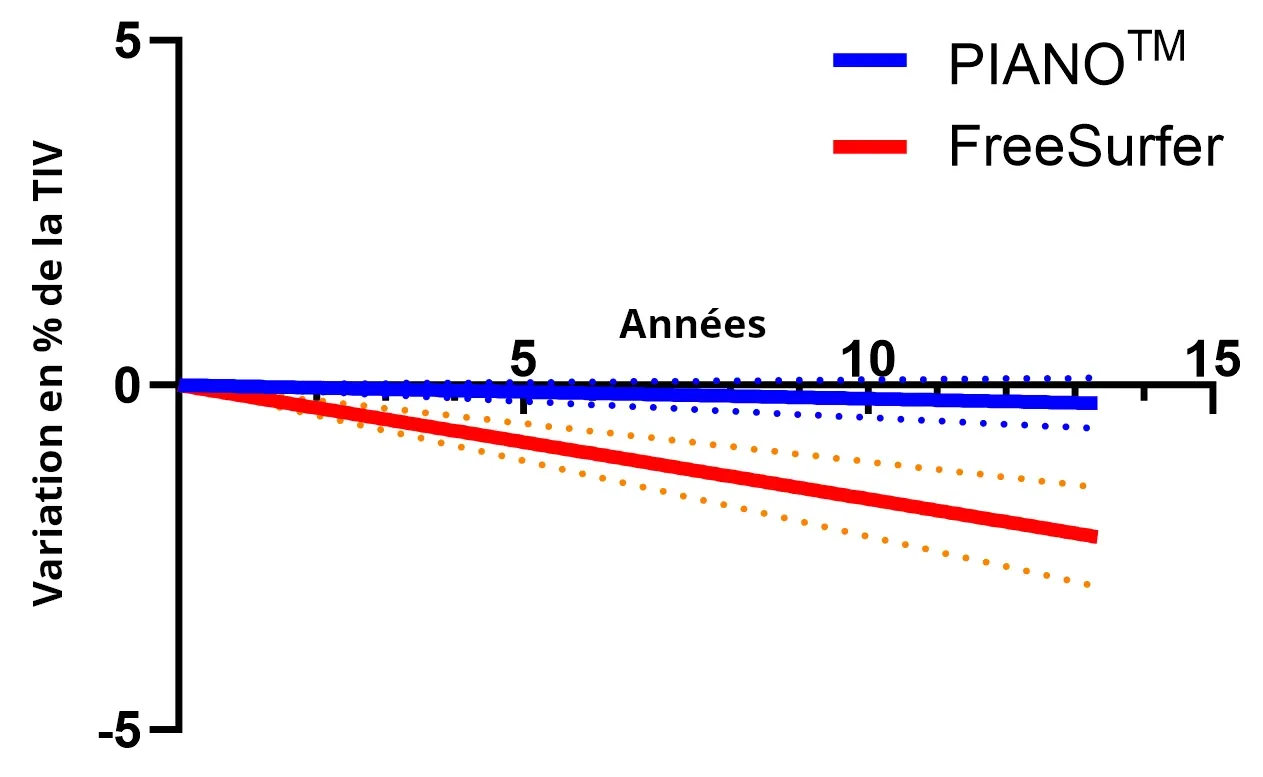
Taux d'échec de segmentation de l'hippocam pe(HC, région rouge rendue en 3D )à l'aide des méthodes de segmentation PIANO™, HippoDeep et FreeSurfer.Les HC gauche et droit représentent le cas où la segmentation des deux côtés échoue simultanément dans le même ensemble de données
Apprentissage profond pour une segmentation robuste de l'hippocampe sur des images IRM
Les modèles d'apprentissage profond, en particulier les réseaux de neurones convolutifs (CNN), ont révolutionné le traitement des images biomédicales en imitant le processus d'identification des caractéristiques du cerveau humain .Les architectures U-Net, avec leurs structures encodeur-décodeur, ont fait progresser les applications de neuroimagerie ,y compris la segmentation des régions d'intérêt (ROI ),en s'entraînant efficacement sur des ensembles de données plus petits augmentés de déformations réalistes (Kruger et al., 2013; Lindsay, 2021 ; Ronneberger et al., 2015)
Nostravaux se concentrent sur la segmentation de l'hippocampe, une tâche difficile en raison de la variabilité de cette région, en particulier dans la maladie d'Alzheimer (MA). À l'aide d'un ensemble de données d'IRM sélectionnées, nous avons entraîné un modèle U-Net avec des données étiquetées, enrichies par un contrôle qualité et des données d'entraînement augmentées. Cette méthode améliore la précision de la segmentation dans les cas difficiles, tels que l'atrophie de l'hippocampe, où les modèles précédents ont échoué (Thyreau et al., 2018)
Notremodèle d'apprentissage profond surpasse les méthodes existantes, telles que FreeSurfer et HippoDeep, en atteignant un taux d'échec de qualité inférieur à 2 % pour chaque hémisphère et < ; 0,4 % pour les deux hémisphères sans corrections manuelles. Cette réduction substantielle du taux d'échec du contrôle qualité a un impact direct sur les coûts des essais cliniques, car le taux d'échec du traitement des données plus faible permet de réduire potentiellement la taille des échantillons des essais. Cette segmentation de l'hippocampe par apprentissage profond a été appliquée à un ensemble de données FTD illustrant les changements précoces rapides au sein de l'hippocampe
En résumé, notre modèle d'apprentissage profond de segmentation de l'hippocampe offre une alternative reproductible et sans biais aux méthodes traditionnelles, répondant aux défis de la neuro-imagerie avec des implications pour des tâches de segmentation plus larges en matière de retour sur investissement.
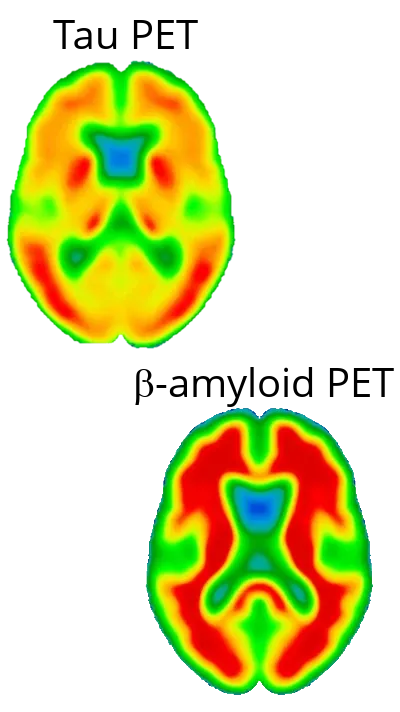
La neurodégénérescence dans la MA est principalement due à la protéine tau, et non à la protéine bêta-amyloïde
Il est de plus en plus reconnu que la neurodégénérescence dans la maladie d'Alzheimer (MA) est principalement due à la pathologie tau, plutôt qu'au dépôt d'amyloïde-β (Aβ). Si les plaques amyloïdes-β ont toujours été considérées comme la marque distinctive de la MA (Gouras, 2015), leur présence n'est pas fortement corrélée à la gravité de la maladie ou à la perte neuronale (Gulisano, 2018). Au contraire, l'accumulation de protéine tau hyperphosphorylée dans les enchevêtrements neurofibrillaires est plus étroitement liée aux schémas de lésions neuronales, de déclin cognitif et de progression de la maladie (Liu, 2020 ; Moore, 2023). La pathologie tau se propage de manière régionale, commençant souvent dans le cortex entorhinal et l'hippocampe avant de progresser vers d'autres régions corticales, en phase avec la progression des symptômes cliniques (Vogels, 2020). Dans nos études, les modèles de propagation et l'analyse de corrélation avec l'épaisseur corticale comme mesure de la neurodégénérescence soutiennent fortement le rôle de la protéine tau dans la neurodégénérescence.
Pour étayer davantage cet argument, les stratégies thérapeutiques qui ciblent la protéine tau, telles que les inhibiteurs de l'agrégation de la protéine tau,les anticorps anti-tau et les kinases de la protéine tau, se sont révélées prometteuses lors des essais précliniques et des premiers essais cliniques (Congdon, 2023).
Image brute de PET harmonisée à la résolution cible commune prédéfinie via un noyau de lissage calculé
Harmonisation de la résolution spatiale des images TEP dans les essais cliniques sans fantôme
SPITFIRE™ de Biospective est une méthode informatique innovante conçue pour harmoniser les images de tomographie par émission de positons (TEP) cérébrale dans le cadre d'études multicentriques (Carbonell, 2024). Les approches traditionnelles s'appuient souvent sur les données fantômes de Hoffman pour estimer la résolution spatiale (Joshi, 2009 ; Harrison, 2020), ce qui peut être limitant en raison de l'indisponibilité des images fantômes et des résultats potentiellement sous-optimaux (Ruwanpathirana, 2024). SPITFIRE™ relève ces défis en estimant la résolution spatiale directement à partir des images TEP elles-mêmes, éliminant ainsi le besoin de données fantômes de substitution. Cette méthode généralise la technique des tracés logarithmiques d'intensité aux trois dimensions, permettant une estimation précise de la résolution spatiale dans les directions axiale et dans le plan. Lorsqu'elle a été appliquée à diverses cohortes, y compris différents traceurs TEP et modèles de scanner, la méthode SPITFIRE™ a permis d'obtenir des estimations de résolution spatiale cohérentes et fiables, avec une variabilité minimale entre les sujets utilisant les mêmes modèles de scanner et paramètres de reconstruction. Cette approche permet non seulement d'améliorer l'harmonisation des images TEP entre lesdifférents centres, mais aussi de fournir un cadre polyvalent applicable à un large éventail de traceurs et de modalités d'imagerie, telles que la TEMP.
Apprenez-en davantage sur l'utilisation de la neuro-imagerie dans les essais cliniques sur la maladie d'Alzheimer et sur nos biomarqueurs d'imagerie.
Découvrez nos autres domaines thérapeutiques
Contenu associé
Informations actualisées sur l'imagerie de la maladie d'Alzheimer et les meilleures pratiques liées à l'évaluation des agents thérapeutiques dans les essais cliniques sur la maladie d'Alzheimer.
Méthode informatique pour l'harmonisation des images TEP du cerveau
Un nouvel algorithme pour normaliser la résolution spatiale des images de tomographie par émission de positons (TEP) du cerveau dans les essais cliniques multicentriques sans fantômes.
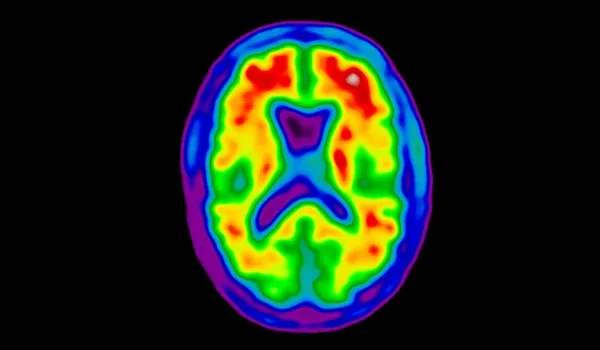
Évolution longitudinale de la TEP Tau dans la MCI et la maladie d'Alzheimer
Vue d'ensemble de l'histoire naturelle de l'évolution de l'absorption et de la fixation du traceur de la TEP Tau dans les troubles cognitifs légers (MCI) et la maladie d'Alzheimer (AD).
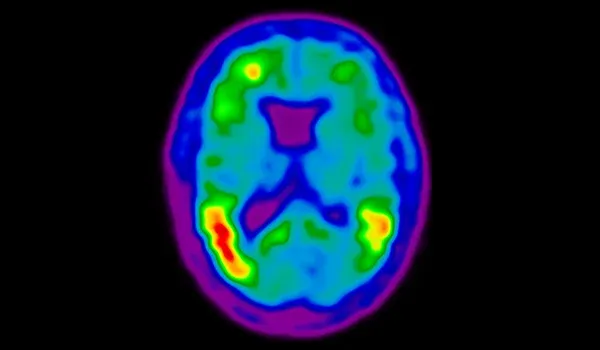
La diminution du métabolisme du glucose cérébral dans la DCL est induite par la protéine Tau
Nous démontrons un rôle plus important de la protéine tau que de la β-amyloïde dans la réduction du métabolisme cérébral du glucose en analysant les images TEP de l'étude ADNI.
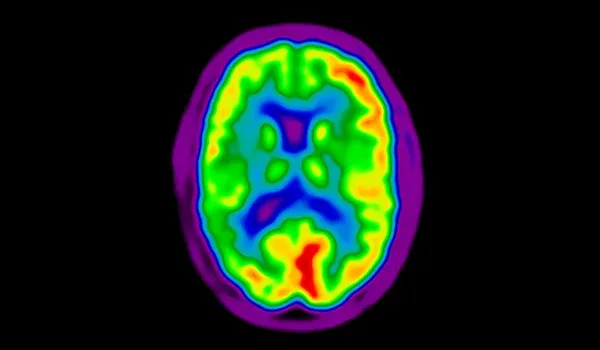
L'association spatiale de l'amyloïde et de la protéine Tau varie en fonction de la cognition
En utilisant une approche informatique, nous avons découvert que la β-amyloïde et la Tau agissent d'une manière spatialement coordonnée qui dépend du stade cognitif de la maladie d'Alzheimer.